-
中药丹参是唇形科鼠尾草属药用植物丹参(Salvia miltiorrhiza Bunge)的干燥根及根茎,具有活血祛瘀、通经止痛、清心除烦、凉血消痈的功效[1]。水溶性的丹酚酸类物质和脂溶性的丹参酮类物质是丹参发挥药效的重要物质基础[2-4]。丹参主要来源于人工栽培,但随着种植规模的扩大,品质退化现象严重[5]。因此,阐明丹参活性成分形成规律对保证其临床用药安全尤为重要。近年来,科研工作者从转录水平对丹参品质形成机制开展了研究,鉴定出多个参与品质调控的转录因子[6-7]。但随着研究的深入,逐渐发现酶的稳定性在药材品质形成中发挥着不可或缺的作用。
泛素-26S蛋白酶体系统通过介导蛋白泛素化降解影响靶蛋白的稳定性[8-11]。其中,F-box蛋白作为Skip-Cullin-F-box(SCF)复合物的核心组分,通过其N端的F-box基序与SKP1相互作用形成骨架,并通过C端的多种蛋白质相互作用结构域选择性识别底物蛋白,这些结构域包括WD 40重复序列、TUB 结构域、Kelch 结构域和富含亮氨酸的重复序列(LRR)等[12]。基于这些不同的结构域,F-box蛋白家族被进一步细分为多个亚家族[13-16]。近年来,F-box-LRR (FBXL)家族蛋白在植物应对生物及非生物逆境中的关键作用日益受到关注。例如,拟南芥茉莉酸受体COI1的C端为18个串联的LRRs结构域[17],能够特异性识别转录抑制子JAZ,并通过26S蛋白酶体途径降解,解除了JAZ对茉莉素途径转录激活因子MYC2的抑制作用,从而激活茉莉素信号途径下游信号防御通路[18-20]。水稻OsCOI1是拟南芥COI1的同源基因,通过与阻遏蛋白JAZ及E3泛素连接酶复合体SCF-COI1的相互作用,诱导一系列防御及生长发育相关基因的表达[21]。拟南芥中的MAX2蛋白,其C端富含LRR结构域,通过调控气孔开合,有效防御丁香假单胞菌和胡萝卜果胶菌的入侵[22]。TIR1/AFBs生长素受体蛋白具有高度保守的F-box-LRR结构域[23],外源病原菌侵染后,miR393过表达导致TIR1水平下降,特异性提高了拟南芥对丁香假单胞杆菌的抗性[24-25]。拟南芥F-box蛋白AFBA1通过脱落酸(ABA)信号途径正调控植物抗旱反应[26]。F-box-Nictaba的表达在拟南芥中受热胁迫、丁香假单胞菌和水杨酸(SA)的诱导, 并通过SA途径介导植物抗病性反应[27]。 拟南芥AT5G15710 基因编码的F-box蛋白参与了对重金属Cu2+或Cd2+的胁迫过程[28]。目前,F-box家族在植物中的功能研究主要集中在拟南芥、水稻等模式植物中,在丹参这一重要药用植物中的研究尚显不足。本研究从丹参基因组中筛选出104条FBXL 基因,并进行生物信息学和表达模式分析,旨在为SmFBXL的后续研究提供依据。
HTML
-
从CNCB网站(https://www.cncb.ac.cn/)中下载丹参基因组(GWHAOSJ00000000)数据及gff注释信息。从拟南芥信息网站(TAIR,https://www.arabidopsis.org/)中下载拟南芥FBXL 基因的氨基酸序列。使用TBtools v2.101中Blast Compare Two Seqs模块,将E值设置为10−5进行本地blast。此外,通过Pfam数据库(http://pfam.xfam.org/)查找丹参FBXL 基因家族特征结构域(PF00646、PF12937、PF13013),通过Simple HMM Search进行丹参FBXL 氨基酸序列的筛选。取两次筛选的序列ID的交集,最后使用在线生物信息学工具国家生物技术信息中心CDD搜索数据库(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)、InterPro数据库(https://www.ebi.ac.uk/interpro/)和SMART网站(https://smart.embl.de), 去除不含F-box和LRRs的序列。
使用ProtParam工具(https://web.expasy.org/protparam/)计算了预测丹参FBXL 基因蛋白的理化特征,包括氨基酸序列长度、理论等电点(pI)、分子量(Mw)、不稳定指数、脂肪族指数和总平均亲水性指数(GRAVY)等指标。使用CELLO在线网站(http://cello.life.nctu.edu.tw/)进行丹参FBXL 基因亚细胞定位预测。
-
从PudMed(https://www.ncbi.nlm.nih.gov/)网站中下载大豆FBXL基因的氨基酸序列,连同丹参和拟南芥FBXL 基因的氨基酸序列进行系统发育关系分析。使用Clustal进行多序列比对,比对完成后使用MEGA11软件Neighbor-Joining(NJ),参数Bootstrap值设为
1000 ,进行系统发育树构建。使用iTOL(https://itol.embl.de/)对进化树进行可视化及美化。 -
使用在线MEME软件(http://www.OMI csclass. com/article/67)分析丹参FBXL 家族成员的基序结构,其中基序的最大数量设置为20,基序的最小宽度设置为6,基序的最大宽度设置为50。从丹参的基因组注释文件中提取丹参FBXL 基因的结构,包括CDS和UTR。最后,使用TBtools v2.101 进行可视化分析。
-
从丹参的基因组注释文件中提取丹参FBXL 基因染色体位置信息,并使用Tbtools软件中Gene Location Visualize from GTF/GFF模块进行可视化。BLASTP程序用于鉴定丹参中的同源FBXL 基因,e值阈值设置为< e−5。使用MCScanX默认参数分析丹参FBXL 基因之间的共线性关系。使用TBtools进行可视化。为了进行共线性分析,从Phytozome v13 网站(https://phytozome-next.jgi.doe.gov/)下载拟南芥基因组序列和注释文件,从CNCB网站南丹参基因组序列和注释文件(GWHASIU00000000)。利用MCScanX程序分析了丹参与拟南芥、南丹参之间的共线性。
-
每个SmFBXL 基因起始密码子(ATG)上游的2 000 bp区域被认为是启动子序列。使用TBtools提取启动子序列并用PlantCARE(https://bioinformatics.PSB.ugent.be/web tools/plant care/html/)预测顺式调控元件。使用TBtools中HeatMap模块对与胁迫反应、植物生长和发育、植物激素响应和光响应的顺式元件进行可视化和总结。
-
丹参植株生长于海军医大学药学系温室,为唇形科植物丹参(Salvia miltiorrhiza Bunge),于丹参生长一月后取其根、叶组织,立即液氮冷冻, 于−80℃冰箱保存,进行转录组测序。使用TBtools的HeatMap模块进行热图可视化,为避免基因表达量差异过大,影响可视化,对数据取Log2(以2为底差异表达倍数的对数)进行标准化,并进行列归一化和聚类分析。
1.1. 丹参FBXL 基因的鉴定、理化性质分析
1.2. 构建系统发育树
1.3. 基因结构和保守基序分析
1.4. 染色体定位、共线性分析和基因复制
1.5. 启动子中顺式作用元件的分析
1.6. 丹参FBXL 不同组织表达模式分析
-
经CDD和Pfam在线分析后最终获得104个SmFBXL序列,均含有F-box和LRRs保守结构域,将所得到的SmFBXL基因序列进行编号,即 SmFBXL1~SmFBXL104。通过ProtParam工具预测理化性质,发现SmFBXL基因长度范围为645 ~ 3 012 bp,编码的蛋白氨基酸长度差异亦较大,为 214~2 362 aa,平均为 494 aa;相对分子质量为 24 333~268 357,与氨基酸长度成正比;等电点为4.41~9.36,其中43个为碱性蛋白质,61个为酸性蛋白质。亚细胞定位预测结果表明,42个 SmFBXLs蛋白定位在细胞核,表明其在细胞核发挥相关调控的功能;9个 SmFBXL蛋白定位在细胞质,表明其在细胞质发挥调控的功能;3 个 SmFBXL 蛋白定位在叶绿体,推测其可能参与叶绿体的基因表达调控;此外,还有1个成员定位于线粒体,37个定位于细胞质膜,12个定位于细胞外间质。
-
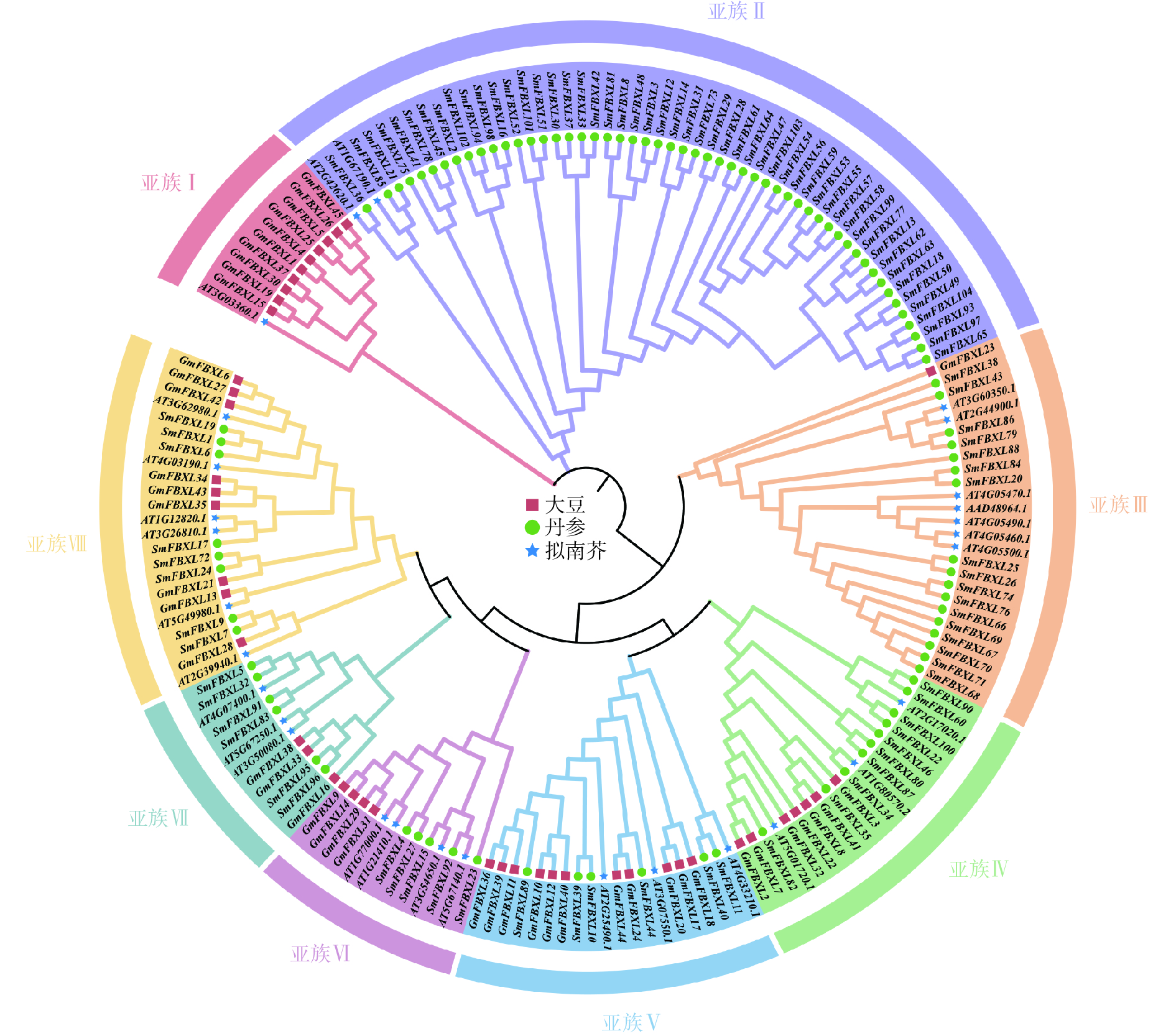
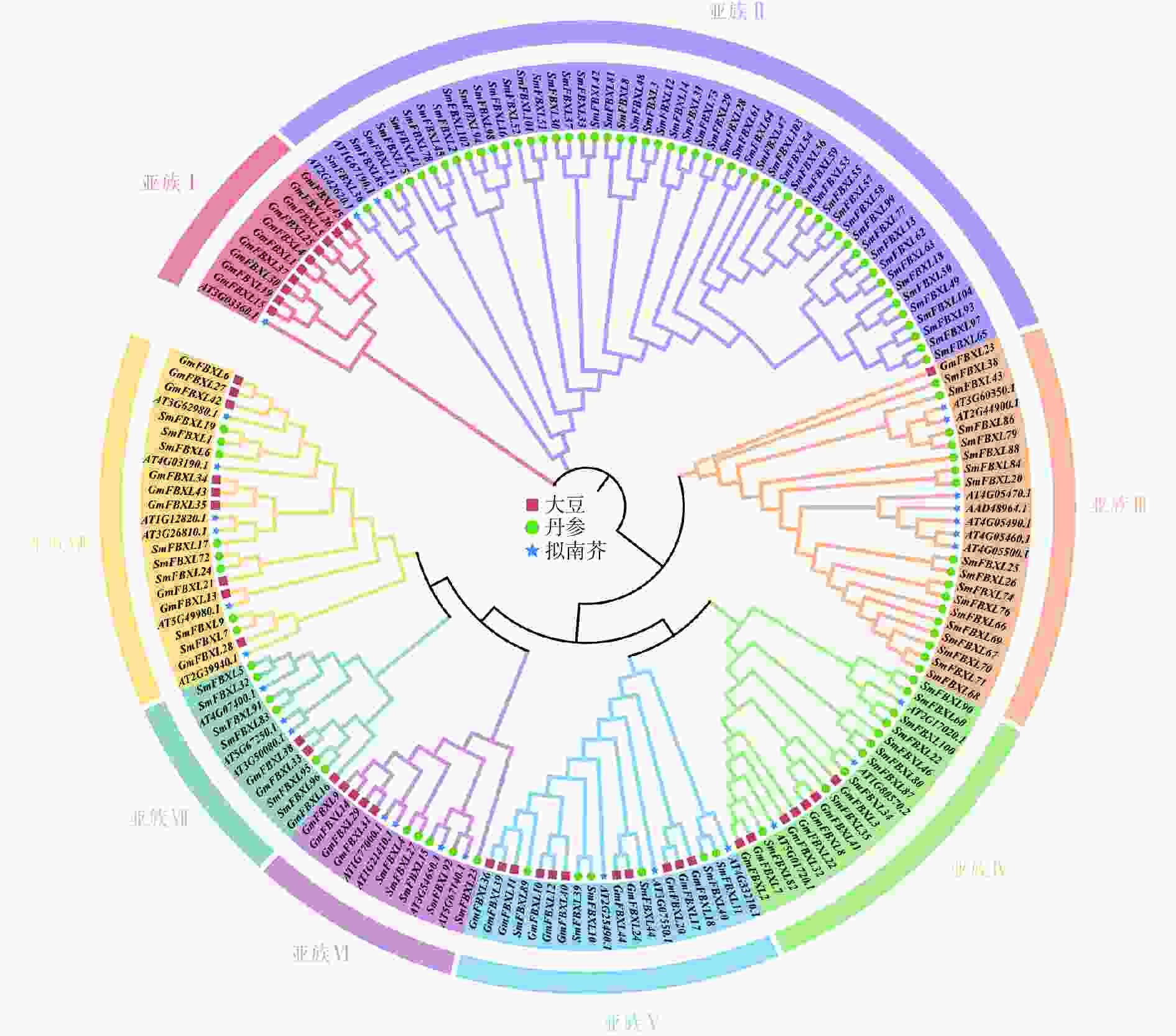
为明确 SmFBXL基因家族成员间的亲缘关系及其与生物学功能的关系,采用NJ法构建丹参、拟南芥和大豆的FBXL 基因家族成员的系统发育进化树(图1)。SmFBXL 基因家族共分为8个亚族(亚族Ⅰ~Ⅷ)。其中,亚族Ⅰ的所有成员大部分来自大豆,仅有一个AtFBXL 家族成员,其他7个亚类中均有SmFBXL家族成员分布。其中,亚族Ⅱ中SmFBXL成员分布最多,有52个SmFBXL成员,其次是亚族Ⅲ、亚族Ⅳ、亚族Ⅷ,分别有17、10、8个,亚族Ⅴ、亚族Ⅶ均有6个SmFBXL成员,亚族Ⅵ仅有5个SmFBXL成员。另外,亚族Ⅱ中仅有SmFBXL和AtFBXL 家族成员,推测SmFBXL在进化过程中可能出现了功能特异蛋白。系统进化上与拟南芥直系同源基因比较相近的FBXL 基因,在其结构与功能上可能比较相似,可以作为推测丹参中FBXL 基因功能的参考依据。
-
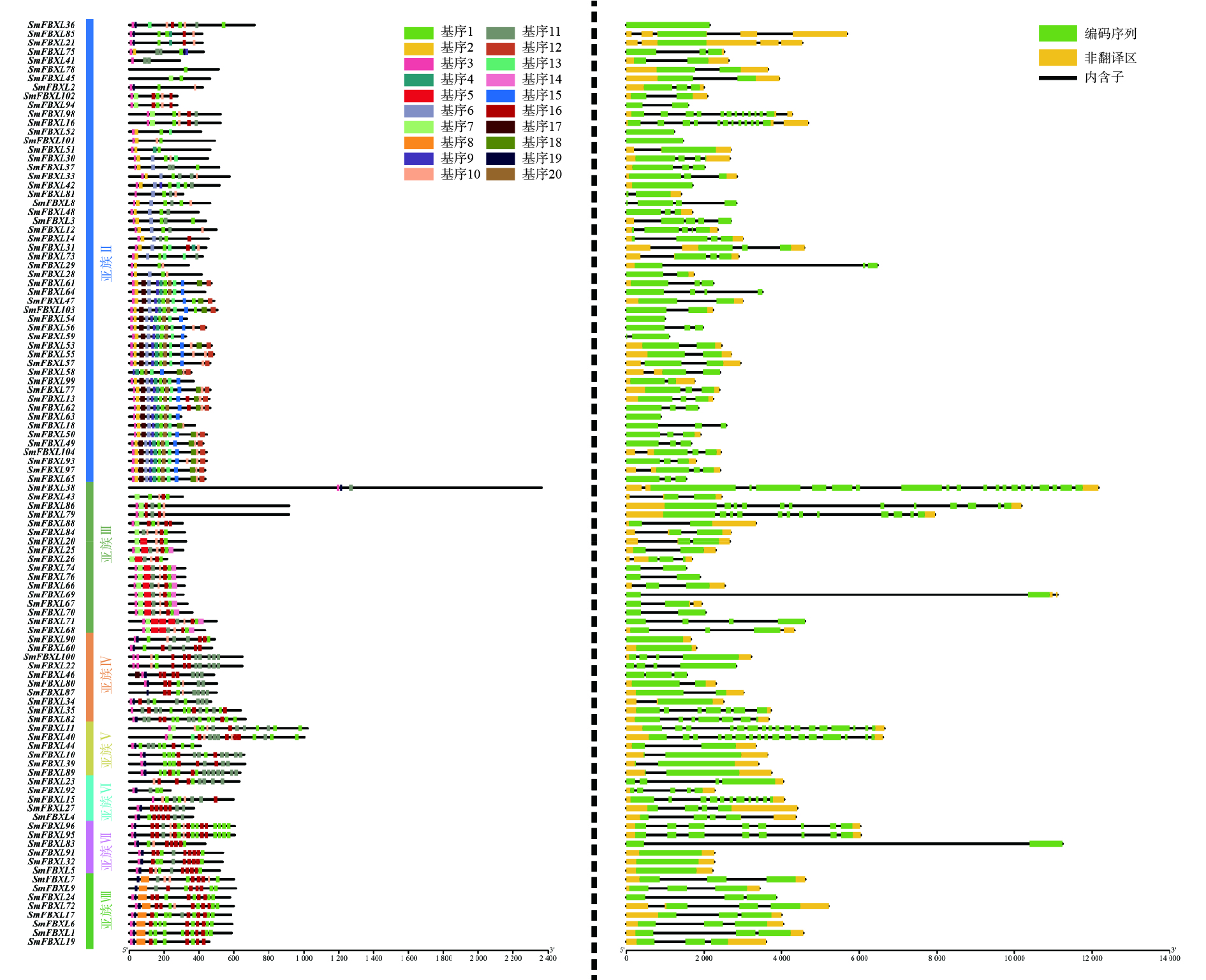
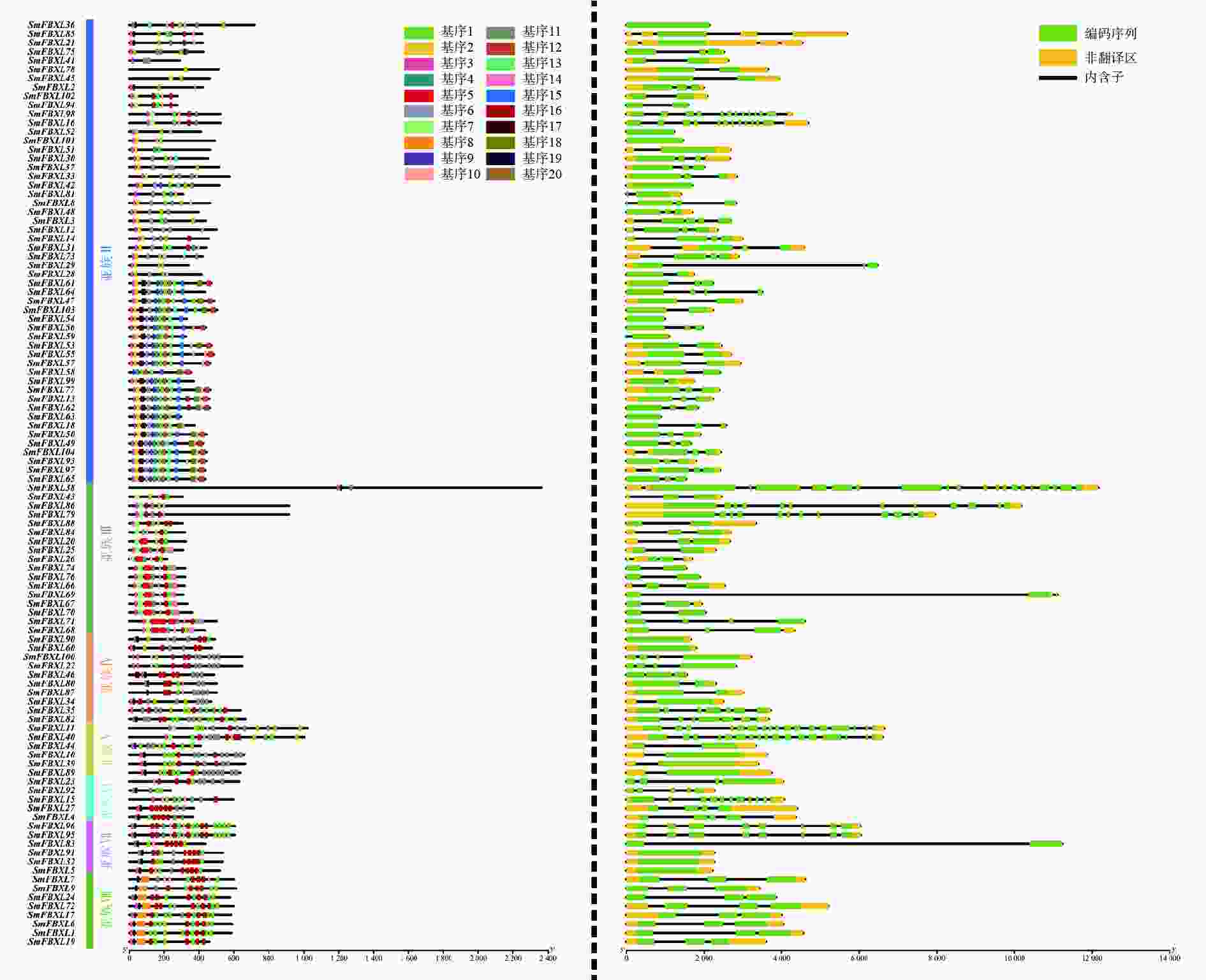
为了进一步分析SmFBXL基因家族成员的结构和潜在功能,利用GSDS、MEME分析SmFBXL基因的外显子与内含子的分布、保守基序。SmFBXL基因家族的蛋白序列中,基序3出现在大多数亚族中,出现频率最高,其与F-Box-LRR保守结构域相关。同一亚族中的大部分SmFBXL基序组成相似,此外,有些基序仅出现在特定亚族中,如亚族Ⅱ成员中含有独有的基序2、基序6,亚族Ⅲ特有的基序5,亚族Ⅷ特有的基序8,推测这些亚族中的FBXL基因可能具有其他重要的生物学功能(图2)。对SmFBXL基因家族成员的结构分析发现,其内含子和外显子数量存在高度的差异性,基本都含有内含子1~10个,多数SmFBXL基因含有2个内含子,同一亚族的SmFBXL转录因子的基因结构相对保守(图2)。
-
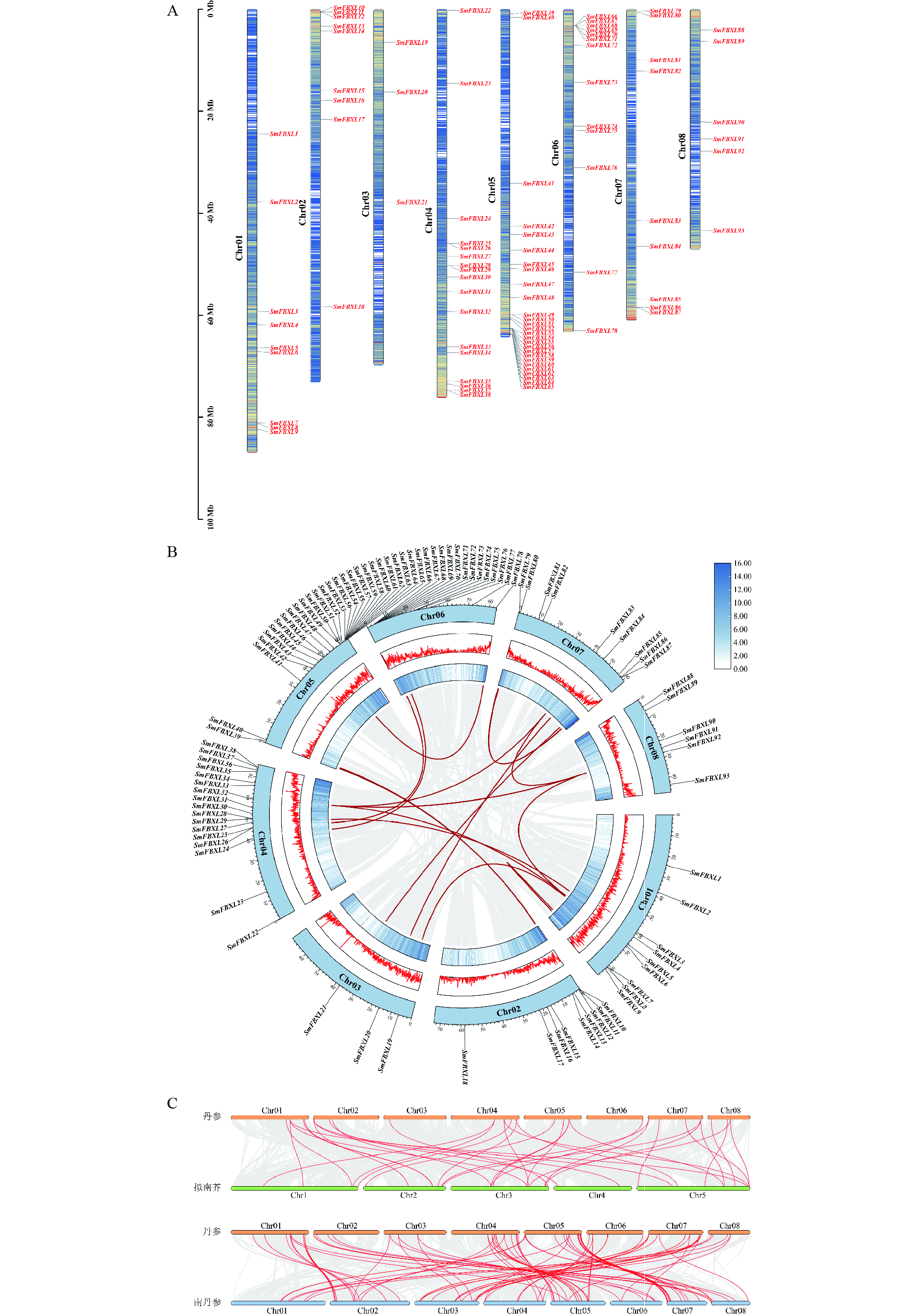
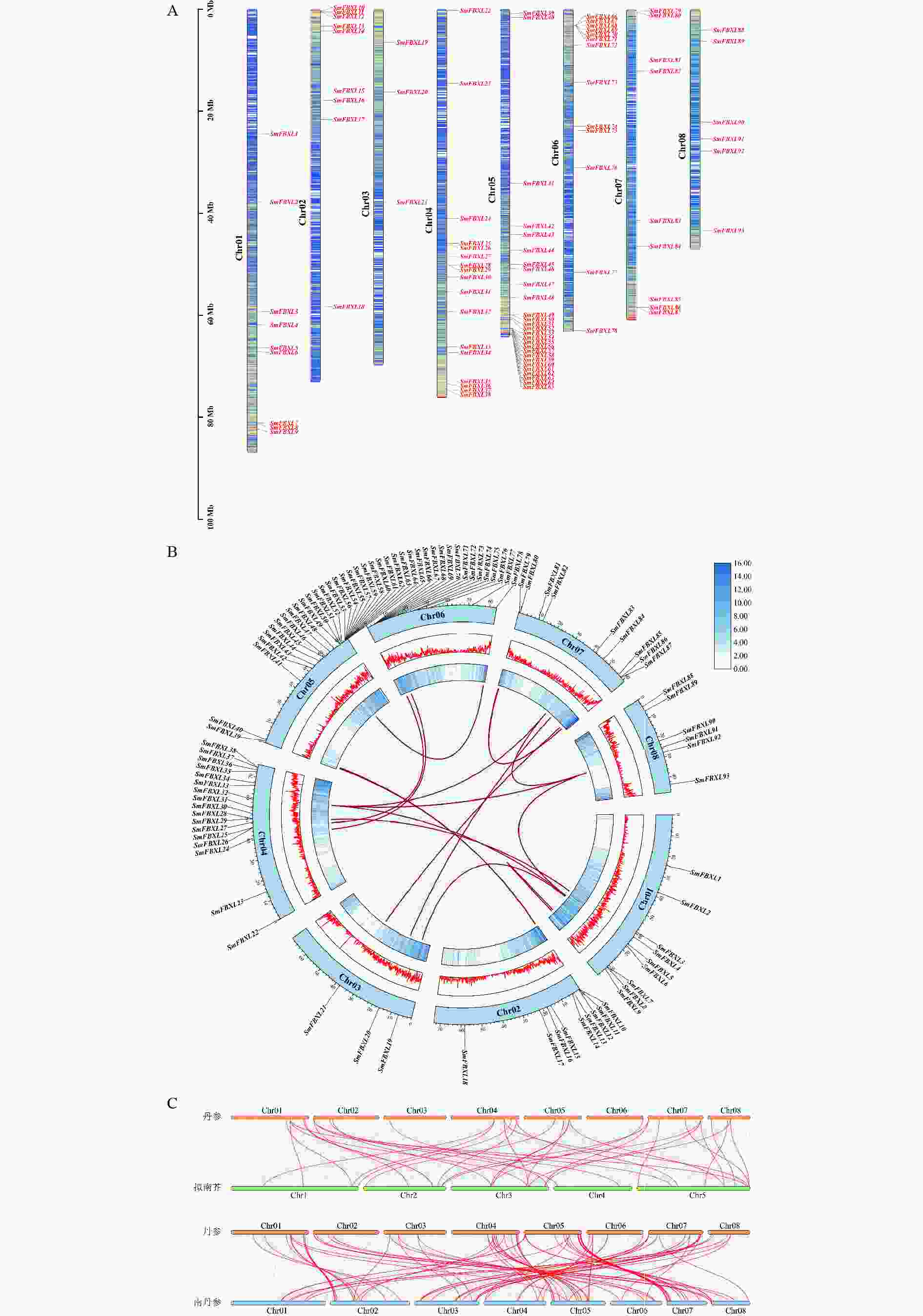
对丹参基因组中获得的SmFBXLs基因进行染色体定位分析(图3A),大部分基因分布在8条染色体的上下臂,每条染色体上的SmFBXL 基因数量为3~15个,基因在Chr01、Chr02、Chr03、Chr06、Chr07、Chr08染色体上分布的较少,集中在Chr04、Chr05染色体的较多,这可能与染色体结构和大小的差异有关。基因家族的形成与扩张很大程度上是由基因复制产生的,为了探究丹参中FBXL 家族的基因扩张情况,本研究对SmFBXL基因家族成员进行了基因复制分析。种内共线性分析(图3B)表明,丹参中FBXL 成员主要通过片段重复进行基因复制,其中涉及到片段重复的基因共有38个,占比36.5%。丹参同拟南芥、南丹参的基因组共线性分析(图3C)表明,丹参中36个FBXL 家族成员同拟南芥FBXL 具有共线性,丹参5号染色体上的基因大多对应拟南芥 3号染色体上;丹参中87个FBXL 家族成员同南丹参FBXL 具有共线性,说明丹参中FBXL 家族成员同南丹参FBXL 具有较高同源性。
-
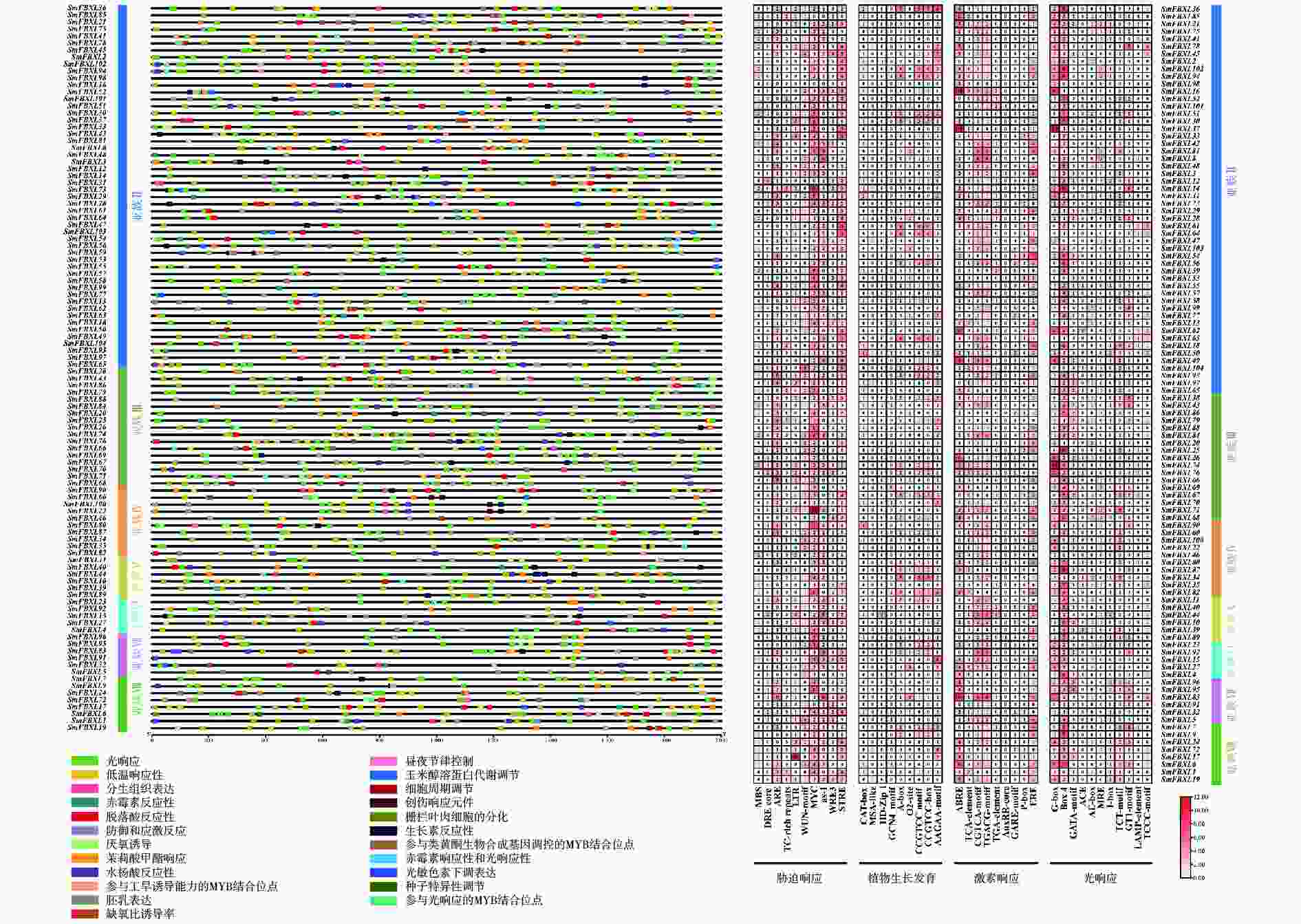
位于基因上游的启动子区域中存在的顺式作用元件可以通过结合转录因子调控基因表达,并对不同环境条件做出反应,这可能为基因功能推测和分化研究提供重要依据。对 SmFBXL 启动子进行顺式作用元件预测,观察到大量不均匀分布在所有基因上的顺式作用元件,将其分为4类:应激响应、光响应、激素响应和生长发育(图4)。统计显示,应激响应类别占比最高,占所有元件的33.7%,包括10种类型的元件,前4个主要元件分别是MYC(低温胁迫)、STRE(渗透压胁迫)、ARE(抗氧化响应)和AS-1(砷胁迫)。第二大类别是光响应元件,占所有元件的26.9%,所有SmFBXL基因家族成员启动子区均含有光响应元件,其中,Box 4的频率最高(光响应元件的34.5%),其次是G-box元件(光响应元件的21.4%)。激素响应占所有元件的25.1%,包括脱落酸(ABA)响应(ABRE)、水杨酸(SA)响应(TCA-element)、茉莉酸甲酯(MeJA)响应(CGTCA-motif和TGACG-motif)、生长素(IAA)响应(TGA-element和AuxRR-core)、赤霉素(GA)响应(P-box和GARE-motif)以及植物雌激素响应(ERE)。ABRE(激素响应元件的26.1%)是最常见的激素响应元件。生长发育元件占比最低(占所有元件的14.3%),包括:负责分生组织表达的CAT-box、CCGTCC-box和CCGTCC motif、负责玉米谷蛋白代谢调控的O2-site、负责次生木质部发育的AAGAA-motif、负责胚乳表达的GCN4-motif、负责细胞周期调节的MSA-like以及负责生长发育的HD-Zip 1等。综上所述,SmFBXL 基因家族的启动子含有低温、渗透压、抗氧化、砷、光响应、SA、ABA、GA、IAA、MeJA和分生组织等响应元件,暗示SmFBXL可能在植物应激防御、激素响应和生长发育中发挥潜在作用。
-
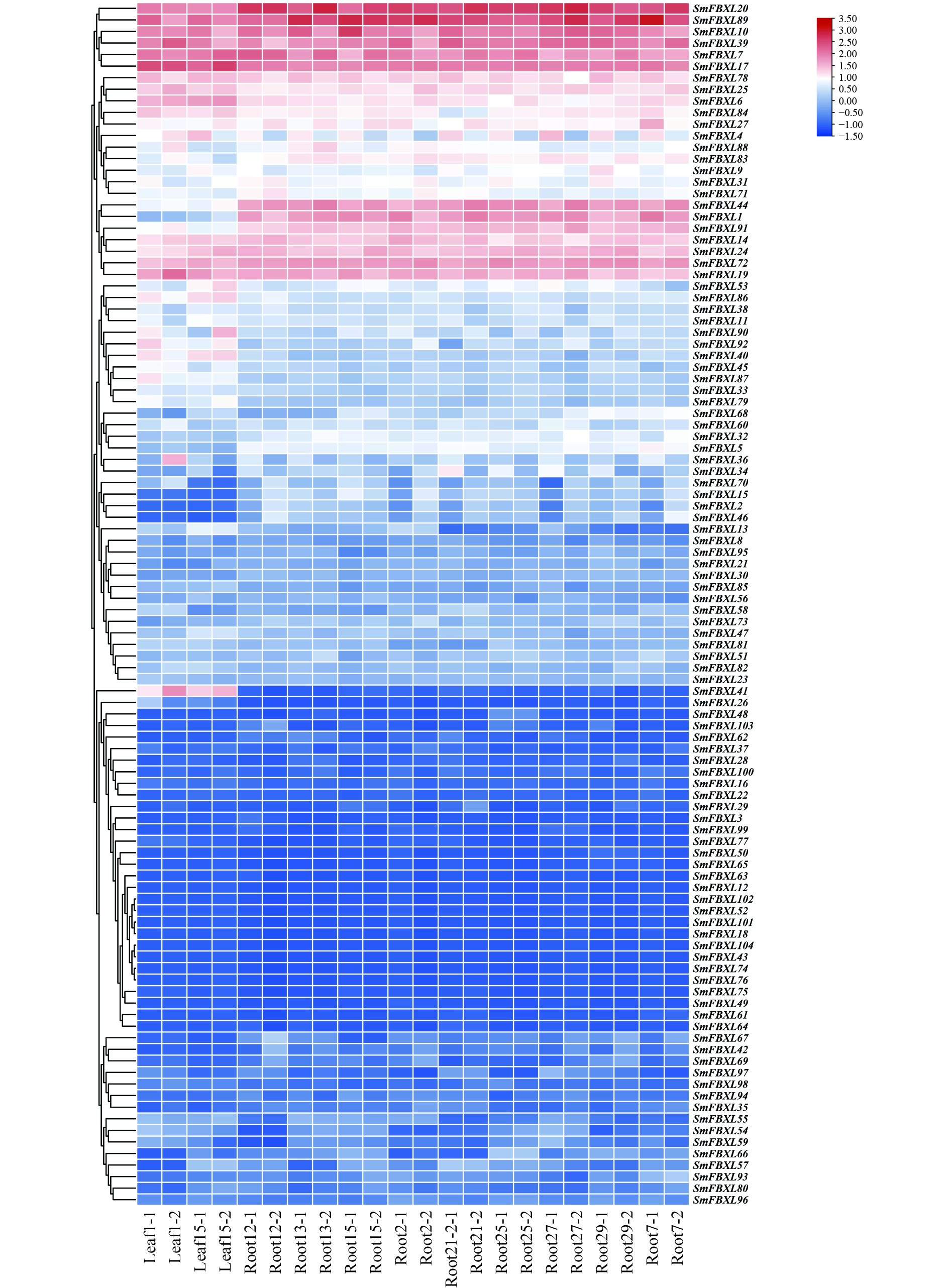
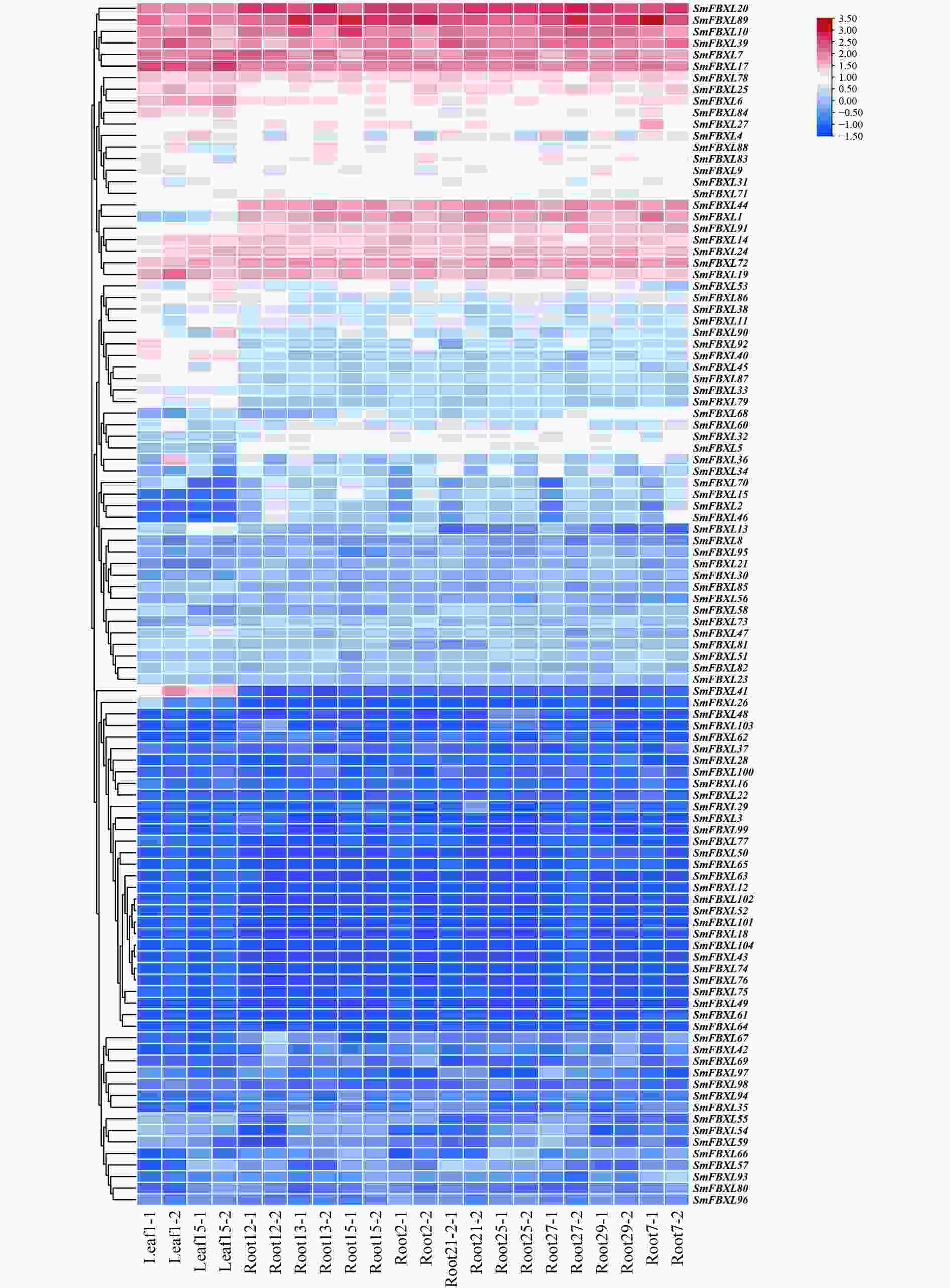
通过转录组测序,分析104个 SmFBXL 基因在不同组织中的表达模式(图5)。结果表明,104个SmFBXL在丹参不同组织中的表达水平差异很大,其中SmFBXL20、SmFBXL10、SmFBXL89、SmFBXL39、SmFBXL7、SmFBXL17、SmFBXL78、SmFBXL25、SmFBXL6、SmFBXL14、SmFBXL24、SmFBXL72、SmFBXL19 这13个基因在叶和根中表达量均相对较高;SmFBXL40、SmFBXL41 2个基因仅在叶片中表达量较高;SmFBXL44、SmFBXL1、SmFBXL91 3个基因仅在根中的表达水平较高;其中44个基因的在叶和根中的表达量都很低,FPKM 值<1。以上结果表明SmFBXL在根和叶中均能发挥功能,其中有13个SmFBXL基因表达量明显高于其他家族基因,可作为后续研究SmFBXL基因家族的候选基因,其原因可能是SmFBXL 基因功能分化所导致的。
2.1. 丹参FBXL 基因的鉴定、理化性质预测
2.2. 丹参FBXL 基因的系统发育分析
2.3. 丹参FBXL基因结构与保守基序分析
2.4. 丹参FBXL 基因的染色体定位、共线性分析和基因复制
2.5. 丹参FBXL 基因启动子中顺式作用元件的分析
2.6. 丹参FBXL 不同组织表达模式分析
-
丹参酮和丹酚酸类物质是丹参中主要生物活性次生代谢产物。因此,明确丹参次生代谢产物的生物合成及其调控机制是丹参质量控制的基础。大量的研究集中在酚酸生物合成的转录调控上,但对控制酚酸生物合成的蛋白质翻译后修饰知之甚少。次生代谢物的产生受环境刺激的影响很大。当植物遭受胁迫时,相应的次生代谢物会迅速合成和积累,同时消耗大量的能量和营养[29],这种生产过剩可能会随着逆境的消除而停止。因此,开启和关闭生物合成途径在植物的生命周期中发挥着同等重要的作用。泛素-蛋白酶体系统作为蛋白质翻译后修饰方式之一,通过介导转录因子或生物合成酶等靶蛋白的降解,在关闭生物合成途径中发挥着重要作用。例如,寒冷胁迫中,花青素可有效清除活性氧造成的氧化损伤,增强耐寒性。低温时,苹果中MdMYB23可激活MdANR的转录,促进花青素的生物合成;常温下,MdBT2介导的UPS可以降解MdMYB23,抑制MdANR,从而终止花青素的生物合成[30],可以看到泛素化修饰在植物抗逆和次生代谢调控中发挥关键作用。
FBXL蛋白通过蛋白-蛋白相互作用域选择性募集靶蛋白,从而控制 SCF泛素连接酶复合物的特异性。FBXL 蛋白分布广泛,迄今为止,在拟南芥、水稻、大豆、玉米、苹果中分别鉴定出29、61、19、16、34 个FBXL 基因[31-33]。然而,关于丹参的FBXL 基因家族的研究还很少,有待进一步挖掘并进行功能鉴定。本研究在丹参基因组中鉴定了104个SmFBXL基因,比多数物种中的FBXL 基因数目多,其大小的差异可能是由于每个物种在进化和分化过程中全基因组多倍体化事件的发生。理化分析表明SmFBXL的氨基酸长度、相对分子质量和等电点差异较大,但基因结构较为保守,基本都含有内含子1~10个,多数SmFBXL基因含有2个内含子,类似的结果亦在水稻[31]、大豆[32]等植物中发现。保守基序对于蛋白质行使生物功能非常重要, 基序3在SmFBXL基因家族出现频率最高,其与F-Box结构域相关,有些基序仅出现在特定亚族中,推测这些亚族中的 FBXL 基因可能具有其他重要的生物学功能。这一现象与油菜[34]的结果相似。SmFBXL 蛋白具有较高的基序保守性,同一亚族的SmFBXL蛋白基序组成基本相似,但也有少数聚在一支的SmFBXL 蛋白基序构成并不相同,推测其可能与SmFBXL 家族成员的功能分化有关。
本研究采用NJ法构建了丹参、拟南芥和大豆的FBXL 基因家族成员的系统发育进化树,将丹参FBXL 基因家族分为了7个亚族。蛋白序列的相似性往往决定其功能的相似性,研究发现拟南芥FBXL 蛋白AT2G42620.1通过调控气孔开合,有效防御丁香假单胞菌和胡萝卜果胶菌的入侵[35],同源进化分析发现,SmFBXL36与AT2G42620.1的同源关系较近,组织表达分析发现在叶中表达量较高,启动子分析发现含有低温响应元件LTR,推测可能与其同源蛋白具有相似的生物学功能。拟南芥AT3G60350.1和AT2G44900.1编码蛋白可以促进侧根发育,与野生型幼苗相比,突变株形成的侧根较少,而高表达株产生的侧根较多[36-37]。系统进化分析发现SmFBXL86、SmFBXL79 与AT3G60350.1、AT2G44900.1同源关系较近,SmFBXL79含有分生组织表达元件CCGTCC 基序和CCGTCC-box,暗示其可能在调控丹参侧根生长中起重要作用。AT5G01720.1(又称作RAE1)与SmFBXL82同属一个分支,是 SCF 型 E3 连接酶复合物的 F-box 蛋白成分。它是明矾诱导调节环路的一部分:STOP1 会诱导 RAE1 的活性,反过来RAE1又会泛素化 STOP1,然后将其降解[38]。AT4G33210编码 SLOMO(SLOW MOTION)蛋白,可以平衡生长素和启动嫩枝分生组织,用于调节温度介导的下胚轴生长[39], SmFBXL40、SmFBXL11和AT4G33210同属一个分支,并且均含有生长素响应元件TGA-element,推测可能具有生长素调控功能。AT3G54650.1和SmFBXL15同源关系较近,可能参与细胞周期基因调控[40]。AT4G07400.1(VFB3)和SmFBXL5、SmFBXL32同源关系较近,与 VFB1、VFB2 和 VFB4 似乎存在功能冗余,当所有 4 个基因的水平都降低时,植株会表现出生长缺陷[41]。综上所述,结合启动子顺式作用元件和组织表达分析,推测SmFBXL家族在丹参逆境胁迫和生长发育中可能发挥重要作用。
基因表达是其发挥生物学功能的前提。SmFBXL基因呈现不同的组织表达特征,104个在丹参不同组织中的表达水平差异很大,部分家族基因仅在叶或根中表达,44个基因在根和叶中表达量都很低。SmFBXL20、SmFBXL10、SmFBXL89、SmFBXL39、SmFBXL7、SmFBXL17、SmFBXL78、SmFBXL25、SmFBXL6、SmFBXL14、SmFBXL24、SmFBXL72、SmFBXL19 这13个基因在叶和根中表达量均相对较高,表明这些SmFBXL可能在不同组织的转录调节中起协助作用,可能是SmFBXL基因功能分化所导致的。部分基因在丹参根中高水平表达,提示这些SmFBXL可能在丹参根发育中发挥重要作用。
当前研究关于SmFBXL基因在丹参中的作用尚待深入探索。该基因家族是否能增强丹参的抗逆性,诱导丹参酮及酚酸生物合成关键酶基因表达,以及其具体作用机制尚不明确。本研究对丹参FBXL家族进行了系统性鉴定,为中药模式植物丹参中FBXL介导的泛素化修饰的生物合成与调控机制解析提供了数据支持,有望为提升丹参的抗逆性及其品质改良提供分子育种上的新策略与参考依据。未来的研究将聚焦于这些核心问题,以期推动丹参种质资源的优化与发展。







 DownLoad:
DownLoad: